Kinase Selectivity 프로파일링 서비스
프로메가의 Kinase selectivity 프로파일링 서비스를 통해 연구자들의 신약 개발 워크플로우를 가속화하세요. 처음부터 끝까지 프로메가 전문가들의 지원을 받을 수 있습니다. 프로메가는 협업적 접근 방식, 혁신적인 기술, 그리고 고품질 표준 관리로 고객의 성공을 위해 최선을 다하고 있습니다.

서비스 개요
프로메가 분석 서비스
Kinase selectivity 프로파일링 서비스는 NanoBRET® Target Engagement Intracellular Kinase Assay를 사용하여 고객의 화합물을 분석하는 서비스입니다. 이 서비스는 HEK293 cell에 transient transfection된 192개 또는 300개의 full-length kinase로 구성된 패널을 사용합니다.
데이터
- 살아있는 세포 내에서의 화합물 affinity와 occupancy의 정량적 측정
- Cellular selectivity 및 off-target activity 측정
- 낮은 에러율로 재현성 높은 ratiometric BRET 결과
- Technical duplicate로 측정된 단일 농도 결과 및 dose response 데이터
서비스 일정
매월 진행하거나 또는 고객 요청에 따라 진행됨
소요 시간
2-3주
프로메가를 선택해야 하는 이유
- 신뢰성 높은 NanoBRET™ Target Engagement 기술
- 처음부터 끝까지 프로메가 전문가들과의 긴밀한 협업
- Cellular assay로 생물학적으로 관련성 높은 결과 제공
기술 개요
How NanoBRET® Target Engagement Works
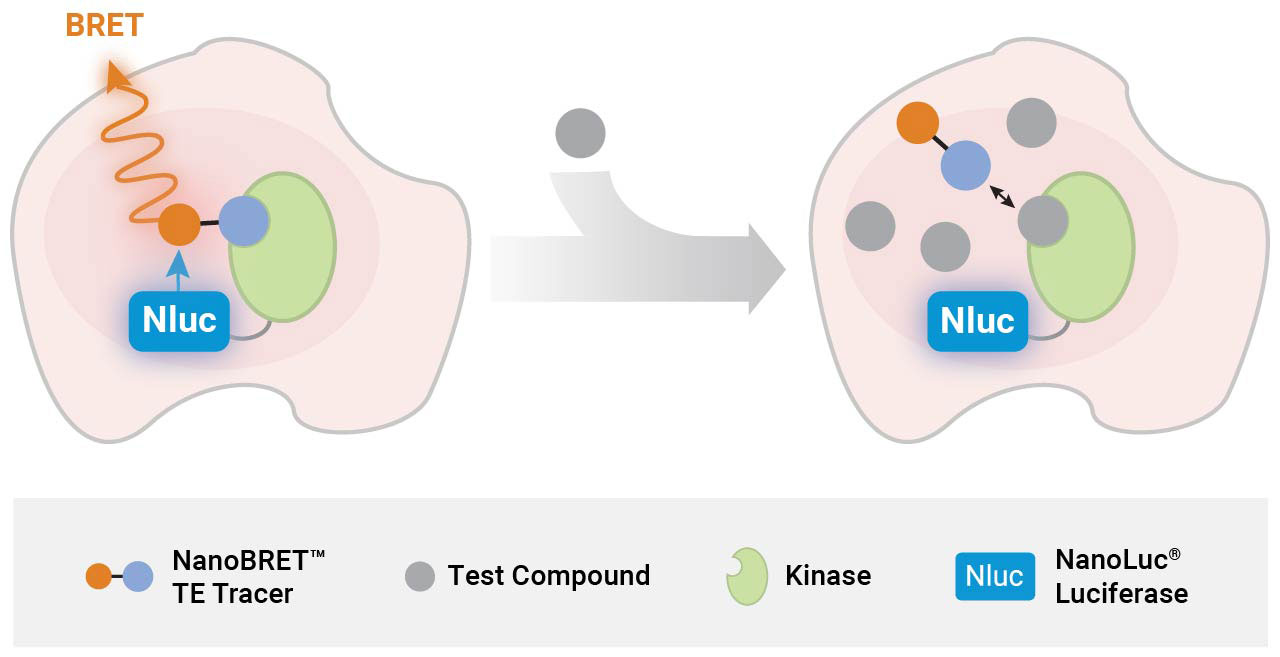
NanoBRET™ TE 기술은 bioluminescence resonance energy transfer (BRET) 방식을 사용하여 타겟 단백질과 화합물의 interaction을 확인할 수 있습니다. 타겟 단백질은 NanoLuc® luciferase와 fusion된 단백질 형태로 세포 내에서 발현시킵니다. BRET signal은 NanoLuc® luciferase의 발광 빛이 타겟-NanoLuc® fusion 단백질에 결합된 NanoBRET® TE tracer에 전달되어 생성됩니다. 세포막을 투과하는 테스트 화합물이 tracer와 경쟁적으로 타겟 단백질에 결합함에 따라 NanoBRET® signal은 감소하게 됩니다. 각 타겟에 대한 최적화된 실험 조건을 사용하여 세포 내 화합물의 affinity와 occupancy를 정량적으로 측정할 수 있습니다. 화합물 occupancy는 BRET signal을 normalization 할 수 있는 control을 사용해 정량화합니다.
Biochemical Kinase Assay 대비 Cellular Kinase Assay의 장점
Cellular kinase assay는 biochemical assay에 비해 실제 세포의 복잡성을 더 잘 반영하는 생리학적 환경을 제공합니다. 이는 약물의 permeability를 평가하고 세포 내의 환경에서 off-target 효과를 확인할 수 있습니다. Biochemical kinase assay는 정확하지만, 세포 내에서의 전반적인 interaction을 고려하지 못하기 때문에 kinase inhibitor를 평가하기 위한 더 통합적인 방법으로 cellular assay가 사용됩니다.
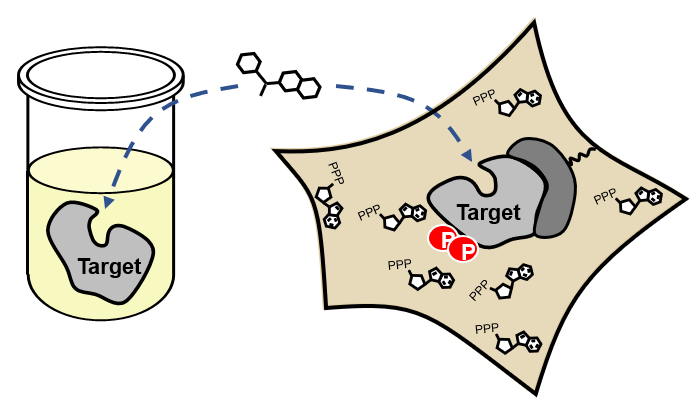
Left: Biochemical kinase assay는 kinase 타겟이 존재하는 간소화된 환경에서 수행됩니다. Right: Cellular kinase assay는 여러 단백질과 interaction이 존재하는 복잡한 세포 환경에서 진행되어 관련성 높은 데이터를 제공합니다.
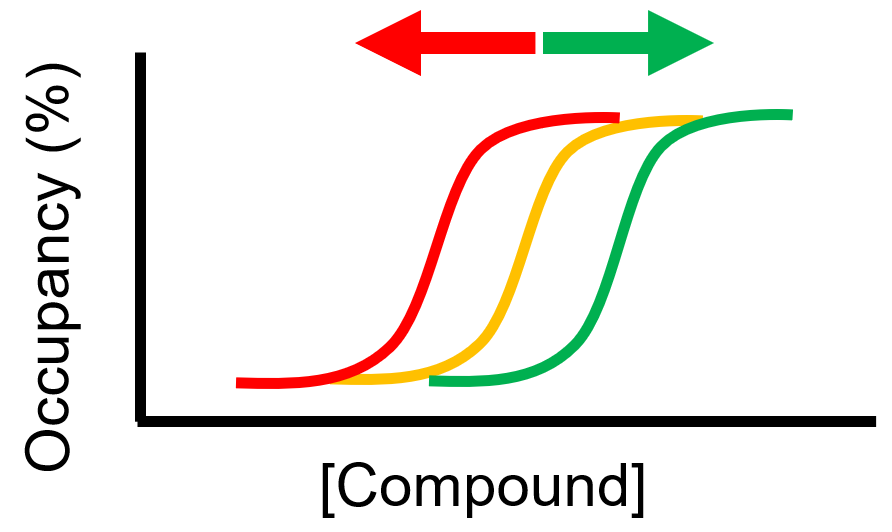
다양한 세포 내의 조건에 의해 occupancy 결과 그래프가 오른쪽 혹은 왼쪽으로 치우치게 됩니다.
- 단백질 complex
- 타겟 활성 상태
- 구획화(compartmentalization)
- 세포막
- 배출(efflux)
- 예측할 수 없는 대사산물(예. Nucleotide)
세포 내에서의 실험 방법으로 Kinase selectivity 프로파일 개선
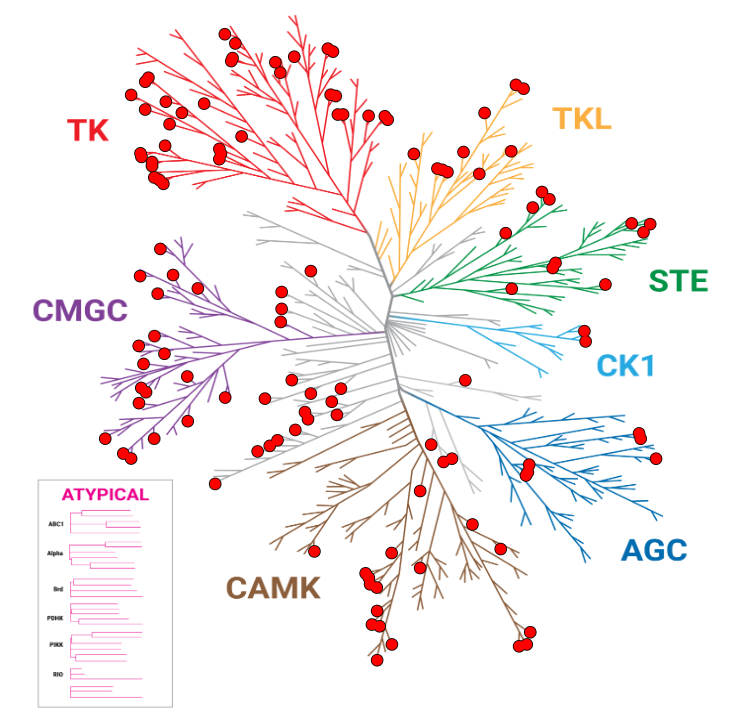
이 dendrogram은 살아있는 세포에서 NanoBRET® TE K300 Kinase Panel을 사용하여 10μM dabrafenib의 타겟 occupancy를 cell-free biochemical 접근법과 비교한 결과를 보여줍니다. Occupancy가 50% 이상인 kinase는 빨간색 점으로 표시되어 있습니다. 실험 결과, dabrafenib은 biochemical 접근법을 사용했을 때 123개의 kinase와 결합한 반면, NanoBRET® TE 방법을 사용했을 때는 50개의 타겟에 결합하여 세포 내에서 더 선택적인 kinase 타겟 프로파일링을 나타냈습니다.
NanoBRET® TE: 50 Hits
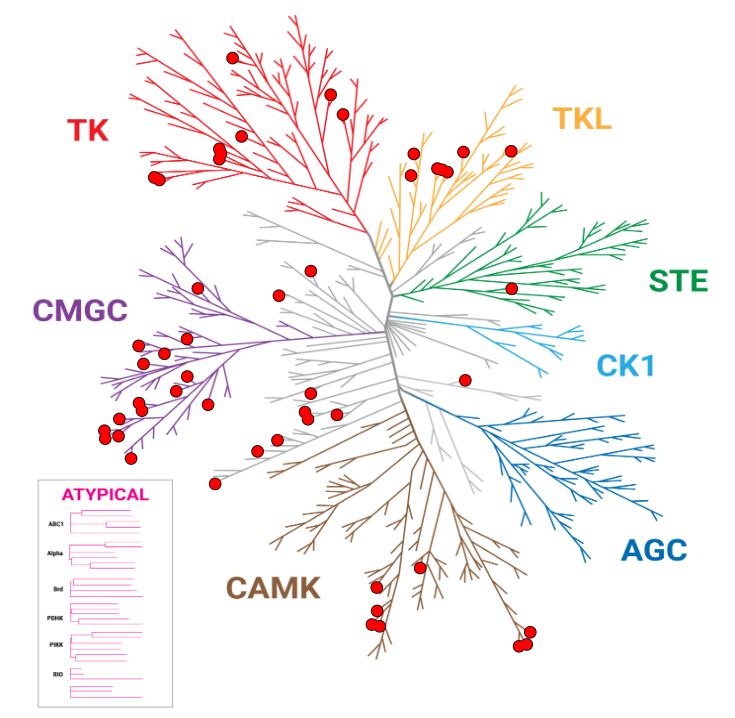
Biochemical: 123 Hits
Kinase Panel
Kinome 전체에 걸쳐 광범위하게 퍼져 있는 kinase가 panel에 포함되어 있습니다. K192 (192개 full-length kinase)와 K300 (300개 full-length kinase) panel에 포함된 전체 kinase 리스트를 확인하시려면 아래를 클릭하세요.
| AAK1 | CDK14 + Cyclin Y | DCLK3 | GSK3B | MAP3K7/TAB1 | NEK9 | PRKCH | STK26 |
| ABL1 | CDK15 + Cyclin Y | DDR1 | HCK | MAP3K9 | NIM1K | PRKCQ | STK3 |
| ABL2 | CDK16 + Cyclin Y | DDR2 | HIPK2 | MAP4K1 | NLK | PRKG2 | STK32A |
| ACVR1 | CDK17 + Cyclin Y | DYRK1A | HIPK3 | MAP4K2 | NTRK1 | PRKX | STK32B |
| ACVR1B | CDK18 + Cyclin Y | DYRK1B | HIPK4 | MAP4K3 | NTRK2 | PTK2 | STK33 |
| ACVRL1 | CDK19 + Cyclin C | DYRK2 | HUNK | MAP4K5 | NTRK3 | PTK2B | STK35 |
| ADK | CDK2 + Cyclin A1 | DYRK3 | ICK | MAPK1 | NUAK1 | PTK6 | STK36 |
| AKT1 | CDK2 + Cyclin E1 | EGFR | IGF1R | MAPK11 | NUAK2 | RET | STK38 |
| AKT2 | CDK20 + Cyclin H | EIF2AK4 k2 domain |
IKBKE | MAPK14 | PAK4 | RIOK2 | STK38L |
| ALK | CDK3 + Cyclin E1 | EPHA1 | INSR | MAPK15 | PAK6 | RIPK1 | STK4 |
| ARAF | CDK4 + Cyclin D3 | EPHA10 | IRAK1 | MAPK3 | PDPK1 | RIPK2 | TBK1 |
| AURKA | CDK5 + CDK5R1 | EPHA2 | IRAK3 | MAPK4 | PHKG1 | RIPK3 | TEC |
| AURKB | CDK6 + Cyclin D1 | EPHA3 | IRAK4 | MAPK6 | PHKG2 | ROCK1 | TEK |
| AURKC | CDK7 | EPHA4 | ITK | MAPK8 | PIK3C3 | ROCK2 | TESK1 |
| AXL | CDK8 + Cyclin C | EPHA5 | JAK2 | MAPK9 | PIK3CA + PIK3R1 | RON | TGFBR1 |
| BLK | CDK9 + Cyclin K | EPHA6 | JAK2 (V617F) | MARK1 | PIK3CB + PIK3R1 | RPS6KA1 | TGFBR2 |
| BMP2K | CDKL1 | EPHA7 | JAK3 | MARK2 | PIK3CD + PIK3R1 | RPS6KA2 | TIE1 |
| BMPR1A | CDKL2 | EPHA8 | JNK3 | MARK3 | PIKFYVE | RPS6KA3 | TLK1 |
| BMX | CDKL3 | EPHB1 | KIT | MARK4 | PIM3 | RPS6KA4 | TLK2 |
| BRAF (V600E) | CDKL5 | EPHB2 | LATS1 | MAST3 | PIP4K2C | RPS6KA6 | TNK1 |
| BRAF | CHEK1 | EPHB3 | LATS2 | MAST4 | PIP5K1B | RPS6KB1 | TNK2 |
| BRSK1 | CHEK2 | EPHB4 | LCK | MELK | PKMYT1 | SBK3 | TNNI3K |
| BRSK2 | CIT | ERN1 | LIMK1 | MERTK | PKN1 | SGK1 | TSSK1B |
| BTK | CLK1 | ERN2 | LIMK2 | MET | PKN2 | SGK2 | TTK |
| CAMK1 | CLK2 | FER | LRRK2 | MKNK2 | PKN3 | SGK3 | TXK |
| CAMK1D | CLK4 | FES | LTK | MLTK | PLK1 | SIK1 | TYK2 |
| CAMK1G | COQ8B | FGFR1 | LYN | MOK | PLK2 | SIK2 | TYRO3 |
| CAMK2A | CRAF | FGFR2 | MAP2K5 | MUSK | PLK3 | SIK3 | ULK1 |
| CAMK2B | CSF1R | FGFR3 | MAP2K6 | MYLK2 | PLK4 | SLK | ULK2 |
| CAMK2D | CSK | FGFR4 | MAP3K10 | MYLK3 | PRKAA1 | SNRK | ULK3 |
| CAMK2G | CSNK1A1L | FGR | MAP3K11 | MYLK4 | PRKAA2 | SRC | VRK2 |
| CASK | CSNK1D | FLT1 | MAP3K12 | NEK1 | PRKACA | SRMS | WEE1 |
| CDK1 + Cyclin B1 | CSNK1E | FLT3 | MAP3K13 | NEK11 | PRKACB | STK10 | WEE2 |
| CDK10 + Cyclin L2 | CSNK1G2 | FRK | MAP3K19 | NEK2 | PRKCA | STK11 | YES1 |
| CDK11A + Cyclin K | CSNK2A1 | FYN | MAP3K2 | NEK3 | PRKCB | STK16 | |
| CDK11B + Cyclin B | CSNK2A2 | GAK | MAP3K21 | NEK4 | PRKCD | STK17A | |
| CDK12 | DAPK2 | GRK7 | MAP3K3 | NEK5 | PRKCE | STK17B | |
| CDK13 | DCLK1 | GSK3A | MAP3K4 | NEK6 | PRKCG | STK24 |

Panel에서 관심 kinase를 찾을 수 없으신가요?
NanoBRET® TE Intracellular Kinase Assay에 사용할 수 있는 kinase vector의 전체 리스트은 Kinase Target Engagement Assay Selection Table에서 확인하실 수 있습니다. 추가적인 kinase를 찾고 계신다면 문의주세요.
Kinase selectivity assay를 직접 진행하고자 하시나요?
한번의 실험에서 192개의 kinase panel에 대해 세포 내 화합물 occupancy를 정량적으로 분석할 수 있는 NanoBRET™ Target Engagement (TE) K192 Kinase Selectivity System을 제공합니다.
결과
화합물의 IC50 결정
프로메가의 서비스는 kinase hit에 대한 화합물 affinity를 결정하는데 도움을 드립니다. 아래 예시 데이터는 selectivity 프로파일링 데이터로부터 확인된 개별 kinase에 대한 IC50 kinase 프로파일을 보여줍니다.

Explore another example:
참고 논문
NanoBRET® TE Kinase Selectivity System은 다양한 peer-reviewed 논문에서 선택된 방법입니다. 자세한 내용은 아래 논문을 참고하세요.
- Vasta, J.D. et al. (2018) Quantitative, Wide-Spectrum Kinase Profiling in Live Cells for Assessing the Effect of Cellular ATP on Target Engagement. Cell Chem. Biol. 25, 206.
- Wells, C. et al. (2020) Quantifying CDK Inhibitor Selectivity in Live Cells. Nat. Commun. 11(1), 2743.
- Ong, L.L. et al. (2020) A High-Throughput BRET Cellular Target Engagement Assay Links Biochemical to Cellular Activity for Bruton’s Tyrosine Kinase. SLAS Discov. 25(2), 176.
- Robers, M.B. et al. (2015) Target Engagement and Drug Residence Time Can Be Observed in Living Cells with BRET. Nat. Commun. 6, 10091
- Jin, H.Y. et al. (2020) High-Throughput Implementation of the NanoBRET Target Engagement Intracellular Kinase Assay to Reveal Differential Compound Engagement by SIK2/3 Isoforms. SLAS Discov. 25(2), 215.
